Inferenza di reti biologiche: CRS4 e Linkalab ai primi posti nella classifica "DREAM5"
 Dentro la complessità delle reti geniche. Prestigioso risultato per due gruppi di ricerca operanti in Sardegna.
Dentro la complessità delle reti geniche. Prestigioso risultato per due gruppi di ricerca operanti in Sardegna.I gruppi di ricerca del CRS4 e del LinkaLab formati da Andrea Pinna, Nicola Soranzo, Vincenzo De Leo e Alberto de la Fuente hanno partecipato alla competizione DREAM5: Dialogue for Reverse Engineering Assessments and Methods organizzata in occasione della terza Conferenza internazionale "Systems Biology, Regulatory Genomics and Reverse Engineering Challenge" alla Columbia University di New York, dal 16 al 20 novembre 2010.
In questa competizione gruppi di ricerca di tutto il mondo partecipano presentando i propri algoritmi. Ai partecipanti vengono forniti i dati di un esperimento del quale devono predire i risultati, noti solo alla giuria. Quest'anno gli organizzatori hanno consegnato i dati di veri esperimenti biologici su batteri e lieviti e uno simulato al computer. Ogni inseme di dati conteneva le misurazioni ottenute in varie situazioni sperimentali: limitando o amplificando l'espressione di un gene, aggiungendo un farmaco oppure introducendo modifiche in una serie temporale.
Alberto de la Fuente, responsabile scientifico del gruppo congiunto CRS4-LinkaLab partecipa da tre anni a questa competizione (si è già classificato nei primi posti nel 2007 e nel 2009) e al CRS4 di Pula (Cagliari) si occupa proprio di sviluppare nuove metodologie per l'analisi integrata dei dati di espressione genica e di genotipizzazione (“systems genetics”) per l'interpretazione delle reti di geni, inoltre lavora all'identificazione delle reti geniche disfunzionali a partire dai dati di espressione genica di soggetti malati (“differential networking”). Spesso le malattie complesse dipendono infatti dall'alterata interazione tra set di geni e non semplicemente da cambiamenti occorsi in un singolo gene. Comprendere le dinamiche di queste interazioni può rappresentare un progresso significativo per la ricerca nel campo delle malattie complesse. Il principio alla base dell'inferenza di reti dalle osservazioni su larga scala - spiega de la Fuente consiste nello stabilire un relazione di causa-effetto tra i geni, utilizzando, per esempio, le perturbazioni di specifici geni e misurando quali di questi cambiano il loro livello di espressione. Questi geni sono legati a quelli perturbati da un rapporto causale. Si distingue poi tra relazioni di causa-effetto dirette e indirette. Il modello di rete risultante, una rete genica, consiste solo di connessioni che corrispondono a relazioni di causa-effetto di tipo diretto tra i geni".
Figura in alto: il poster che illustra il lavoro del team ALF Elucidating Transcriptional Regulatory Networks from heterogeneous gene-expression compendia.
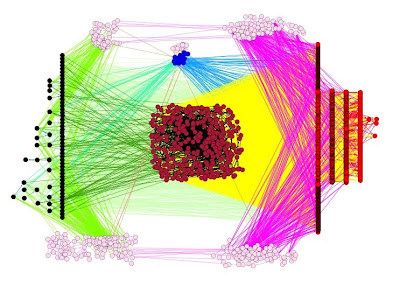
Figura in basso (creata da Gianmaria Mancosu con il software Pajek): la configurazione 'bow-tie' della rete genica del lievito ottenuta dal gruppo di Bioinformatica del CRS4 dall'integrazione delle analisi di espressione genetica e dai dati genotipici
Bioinformatics Laboratory (CRS4)



Commenti